P
 x
x 
I. hederacea (coH, Cse, tw?) 931 (f-1,2,3 p B ca m v-1 bv, tw+)
↓
F1 
アサガオの分子マーカーを用いた連鎖地図の作製
今後アサガオを使って、遺伝学的・分子生物学的な解析を行っていく上で詳細な連鎖地図は欠かせない。近年のゲノム生物学の発達に伴い、連鎖地図作製に向けてさまざまなテクニックが考案されており、アサガオでも同じようなテクニックを用いて詳細な連鎖地図を作製している。そのため、様々な植物で利用されている、PCR法を応用した分子マーカーをできるだけ密に作製する。そして古典地図で使われている表現型マーカーと分子マーカーの両方を乗せた連鎖地図を作製する。
96年夏、アサガオと交配可能な近縁種であるアメリカアサガオ(I. hederacea)を除雄して、Q931系統の花粉をかける。交配成功率は5〜10%とかなり低い。
 x
x 
↓
F1 
97年夏。アメリカアサガオのくびれのある葉の表現型(ヘデラセア葉)が優性であることがわかる。またアサガオは花筒が着色しているが、ヘデラセアは着色していない(tw遺伝子?)が、F1では花筒が着色している。これを自殖させてできるだけ多数の種子を採種した。
98年夏。F2系統を225個体展開し、分離してきた表現型マーカーの観察・記録、DNA抽出用サンプルの採種を行った。またアメリカアサガオは非常に早咲きであり、その形質に関しても早咲きの個体が分離してくるため各個体の開花時期を記録した。
F3 
F2系統で劣性形質をヘテロに持っているか(+/m)、持っていないか(+/+)はマッピングの精度を上げるために重要な情報である。ヘテロ形質をチェックするために98年から99年の冬にかけて温室でF3を栽培し、分離してきた表現型を観察した。たとえば、F3で白い花を咲かせる個体が分離してきた場合、F2個体はca/+の遺伝子型だったと考えられる。
2)AFLP法によるF2個体のマーカー分析
アサガオの連鎖地図をPCR法ベースの分子マーカーを用いて作製する。まず15の連関群のおおよその地図を作るため、PCR法をつかった分子マーカーで、一度に多数のバンドが得られるRAPD(Random Amplifeid Polymorphic DNA)法を用いてみた。この方法は10塩基のランダムプライマーを用いて増幅してくるバンドの多型に基づいている。OPERON社が市販しているプライマーのAセットおよびBセットを用いて、アサガオとアメリカアサガオ、これらのF1の3サンプルのDNAを反応させた。
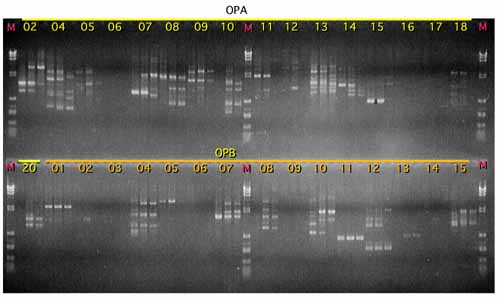
その結果、ほとんどのマーカーで、アサガオとアメリカアサガオで差のあるバンドが検出されたが、再現性の面で問題があった。
そのため、AFLP(Amplified Fragment Length Polymorphism)法を用いることにした。これはRFLP(Restriction Fragment Length Polymorphism)法と類似の方法であるが、EcoRIとMseIで切断した断片の数を減らす工夫がしていある。このAFLPマーカーとRAPDマーカーはこれらはバンドが増幅することに関して、ホモ個体(両親由来のそれぞれからバンドが増幅する)とヘテロ個体(片方の染色体からしかバンドが増幅していない)に関して区別がつかない優性マーカー(dominant marker)のため、マッピングの精度の面で問題があるが、一度に多数のマーカーが作れるという利点がある。
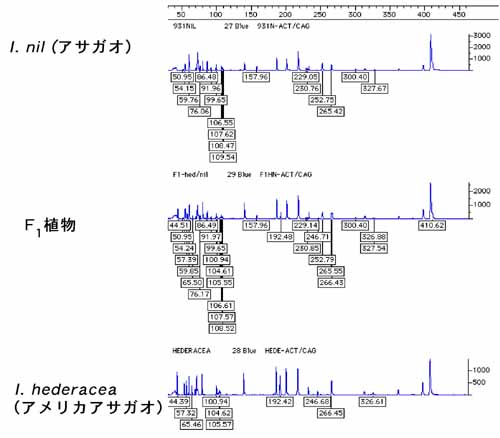
これはアサガオとアメリカアサガオ、これらのF1の3サンプルのDNAをPerkin-Elmer社のPlant
Mapping Kitを用いて反応し、同社のABI-Prizm 310 Genetic
Analyzerにかけたサンプルである。この中で線で囲んだ数字はアサガオとアメリカアサガオの間で差のあるバンドを示している。このようにF1個体では両親のバンドが両方出ている。このF1植物を自殖させ、分離してきたF2ではこれらのバンドの有る無しも分離してくる。
アサガオとアメリカアサガオで差があり、はっきり読めるピーク(バンド)をAFLPマーカーとした。このサンプルのようにEcoRI(蛍光あり)プライマーがACTで、MseI(なし)がCAGの場合で192.42bpのピークがアメリカアサガオで見られた場合、正式なマーカー名はCTAG192.42h としマッピング用の省略名はCTAG09hのように名前をつけた。同様の反応をF2個体、225個体のうち、系統番号(#1〜#178)に行い、ホモ・ヘテロ接合体は判別が難しいのでバンドの有無のみを目で確認し、01データに変換した後、アサガオ由来のnマーカーでバンドが出ないもの(0/0)はアメリカアサガオ側の対立遺伝子をホモ接合で持っていると考えられるためB、バンドが出るものはアサガオ側の対立遺伝子をホモ接合またはヘテロ接合で持っている(1/1or 1/0)と考えられるためDに変換した。hマーカーも同様に、A、Cに変換した。現在までにアメリカアサガオとの間で多型を示す400以上のマーカーを得ている。
これらのデータをマッピング解析ソフトMapmaker 3.0b (MS-DOS version, White head institute; MIT)で解析した。まずマーカーを連鎖群に分類し、それぞれのマーカー間の組換率に基づき、連鎖地図を解析した。LOD(Log Odds)スコアを上げていくと複数の連鎖群に別れるが、15群に多数のマーカーが分類された。このことは染色体数から予測される連鎖群の数と一致していた。
現在、連鎖地図はおおよその形はできているが、分離比のおかしいもの、ミスタイプのもの等を除いて校正する作業を行っており、公開はしていない。
3)すでにクローン化された遺伝子の多型マーカーの作製・解析
dp (duplicated、牡丹)はC-classのMADS-box遺伝子であり、今井によって第6染色体の端(0.0)にマップされている。アメリカアサガオとの間で第3イントロンの長さに多型があったためこれを判別するプライマーを設計し、F2個体の遺伝子型を判定しマッピングに用いた。
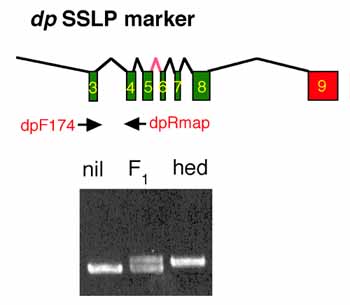
このようなSSLP(Simple Sequence Length Polymorphism)マーカーはAFLPマーカーとは異なり、共優性マーカー(codominant maker)と呼ばれ、ホモ個体とヘテロ個体の区別ができるためマッピングの精度を上げることができる。
ほかにも牡丹と同じC-class(C機能)MADS-box遺伝子である芍薬(pn; peony)についてもSSLPマーカーを作製し、解析を完了している。
4)今後の展望
AFLPマーカーはこのようにおおよその連鎖地図を作製するのには有効であるが、優性マーカーのため精度の面で共優性マーカーより劣っている。今後クローン化された遺伝子やEST(Expression Sequence Tag)クローンについて、共優性マーカーであり、より信頼性の高い、SSLPマーカーや制限酵素部位を挟むようなCAPS(Cleaved Amplifeid polymorphic sequence)マーカーによる連鎖地図に切り替えていく予定である。また古典マップと一致させるために今回用いていない表現型マーカーを持つ系統とアメリカアサガオを交配した系統を用いて対応づけを行う。今回は1(v, B), 2(coH), 3(m), 6(dp), 8(p)に関して古典マップと分子マーカーを用いたマップを一致させることができたが、4, 5, 7, 9, 10は未対応である。4に関してはmg(暗紅)ac(南天)、5はct(渦)、7はre(洲浜)、9はdl(笹)、10はcs(石畳)が他に類似の突然変異がなく適当だと考えている。また現在15群のうち10群にしか表現型マーカーが乗っていないが、全ての連関群に表現型マーカーを位置づけることも目標としている。
他の問題点として、交配親に用いているアメリカアサガオとアサガオの交配成功率が非常に低い(5〜10%)ということが挙げられる。クローニングして、分子マーカーを作製した場合は問題にならないが、新規に突然変異体を単離して連鎖群を決める場合、この点が問題となる。そのため普通に交配できて多型もアメリカアサガオに次いで多い、アフリカ系統のアサガオを交配親に使うことも考えている。しかしアフリカ系統は以下のような重大な短所がある。短日条件にたいして鈍くなっており、開花が非常に遅いため自然条件では採種できにくい。アサガオの突然変異系統との交配種も同様に開花が非常に遅い。またアフリカ系はアサガオと比べて非常に大型なので栽培面積を要し、栽培管理が大変である。